云南野桑蚕线粒体基因组特征及系统发育分析
Mitochondrial Genome Characteristics and Phylogenetic Analysis of Yunnan Bombyx mandarina
-
Keywords:
- Bombyx mandarina /
- mitogenome /
- phylogenetic analysis
-
水环境是水生生物的生存环境,良好的水质是水生生物健康生存的基础,水质变化导致的应激不仅会影响水生动物,同时也会影响病原菌,增加疾病发生的风险。疾病的发生是环境—病原—宿主间相互作用的结果,目前已有大量研究报道了水生动物在温度、pH、氨氮与亚硝酸盐等环境因子胁迫下对病原的易感性增强[1-2],也有研究表明环境因子如温度、渗透压、pH、活性氧、氮化合物和无机离子浓度等因子对细菌毒力也有明显影响[3],如18 ℃时香鱼假单胞菌(Pseudomonas plecoglossicida)对大黄鱼的致病性强于25 ℃,此时细菌T6SS和铁摄取系统相关基因的表达较25 ℃显著上调[4];嗜水气单胞菌(Aeromonas hydrophila)毒力基因溶血素(AHH)、气溶素(AerA)、外膜蛋白(OMP)和黏附素(Aha)在15 ℃与pH 7.0时高效表达,但在4 ℃与pH 9.0时则受到抑制[5]。pH不仅影响细菌毒力基因的表达,也会对细菌增殖、生物膜形成和胞外产物(extracellular products,ECPs)活性与致病性产生重要影响。研究表明:嗜水气单胞菌最适生存pH值为6.5[6],当环境pH值低于4时不能形成生物膜[7],pH 7.5时其分泌的ECPs酶活性最强,且对对虾和鲫鱼等有较强的致病性[8]。
拟态弧菌(Vibrio mimicus)广泛存在于水环境中,可感染鳌虾、蟹、龟和多种鱼类,尤其对以黄颡鱼(Pelteobagrus fulvidraco)和南方鲇(Silurus meridionalis)等为代表的淡水鲇形目鱼类危害最为严重[9]。目前对于该菌的主要毒力因子种类、胞外产物和基因组特性[10]、感染鱼类的病理损伤特点及病原的分布规律[11]等进行了较为深入的研究,这些研究为阐释拟态弧菌的致病机制提供了重要的支撑。pH是一项重要的水质指标,其变化对拟态弧菌感染与致病相关特性的影响尚不清楚,因此,本研究在水生动物正常生长的pH范围 (6.0~9.0)内,研究不同pH对拟态弧菌感染相关表型及胞外产物特性的影响,以期为进一步揭示其感染与致病机制提供依据,同时也为养殖生产中通过调节水体pH来防控拟态弧菌病提供参考。
1. 材料与方法
1.1 菌种与试验动物
拟态弧菌SCCF01株由四川农业大学鱼病研究中心保存;供试斑马鱼购自成都某斑马鱼养殖基地,体长(2.95±0.20) cm。
1.2 主要试剂
LB培养基、琼脂、卵黄琼脂和明胶购自青岛海博生物有限公司;M199培养基购自HyClone;BCA蛋白浓度检测试剂盒购自北京索莱宝生物科技有限公司;CCK-8细胞活性检测试剂购自上海生工生物科技有限公司。
1.3 pH对拟态弧菌感染相关表型的影响
1.3.1 对增殖的影响
采用吸光光度法测定pH对拟态弧菌增殖的影响。分别取新鲜拟态弧菌菌液(OD600=1.0) 500 μL接种于50 mL不同pH (6.0、7.0、8.0、8.5和9.0)的LB肉汤(n=3),振荡培养,每2 h测量OD600值,连续测量24 h,绘制生长曲线。
1.3.2 对泳动的影响
分别取新鲜拟态弧菌菌液0.5 μL接种于不同pH (6.0、7.0、8.0、8.5和9.0)且含0.3%琼脂的LB半固体培养基(n=3),28 ℃静置培养48 h后测量菌圈直径。
1.3.3 对自聚集的影响
采用吸光光度法检测菌株的自聚集。分别取新鲜拟态弧菌菌液100 μL接种至10 mL不同pH (6.0、7.0、8.0、8.5和9.0)的LB肉汤(n=3),28 ℃静置培养48 h,测定上清液OD600值,并计算自聚集率。自聚集率=[1-(At/A0)]×100%,式中:At为特定时间的吸光度;A0为初始吸光度[12]。
1.3.4 对生物膜形成的影响
参考LUO等[13]的方法,采用微孔板法检测生物膜形成。分别取新鲜拟态弧菌菌液10 μL加入不同pH (6.0、7.0、8.0、8.5和9.0)的190 μL LB肉汤(n=3),静置培养48 h,经结晶紫染色、无菌磷酸缓冲液(phosphate buffered saline,PBS)清洗和醋酸溶解后测定OD570值。
1.4 pH对拟态弧菌胞外产物(ECPs)特性的影响
1.4.1 对ECPs蛋白质量浓度的影响
分别取新鲜拟态弧菌菌液(OD600=1.0) 4 mL 接种至不同pH (6.0、7.0、8.0、8.5和9.0)的400 mL LB肉汤(n=3),28 ℃、180 r/min振荡培养48 h后调整各组拟态弧菌菌液OD600值为1.0;采用饱和硫酸铵法[14]提取拟态弧菌 ECPs,并采用PBS定容至1 mL,通过BCA蛋白浓度检测试剂盒测量其蛋白质量浓度。
1.4.2 对 ECPs酶活性的影响
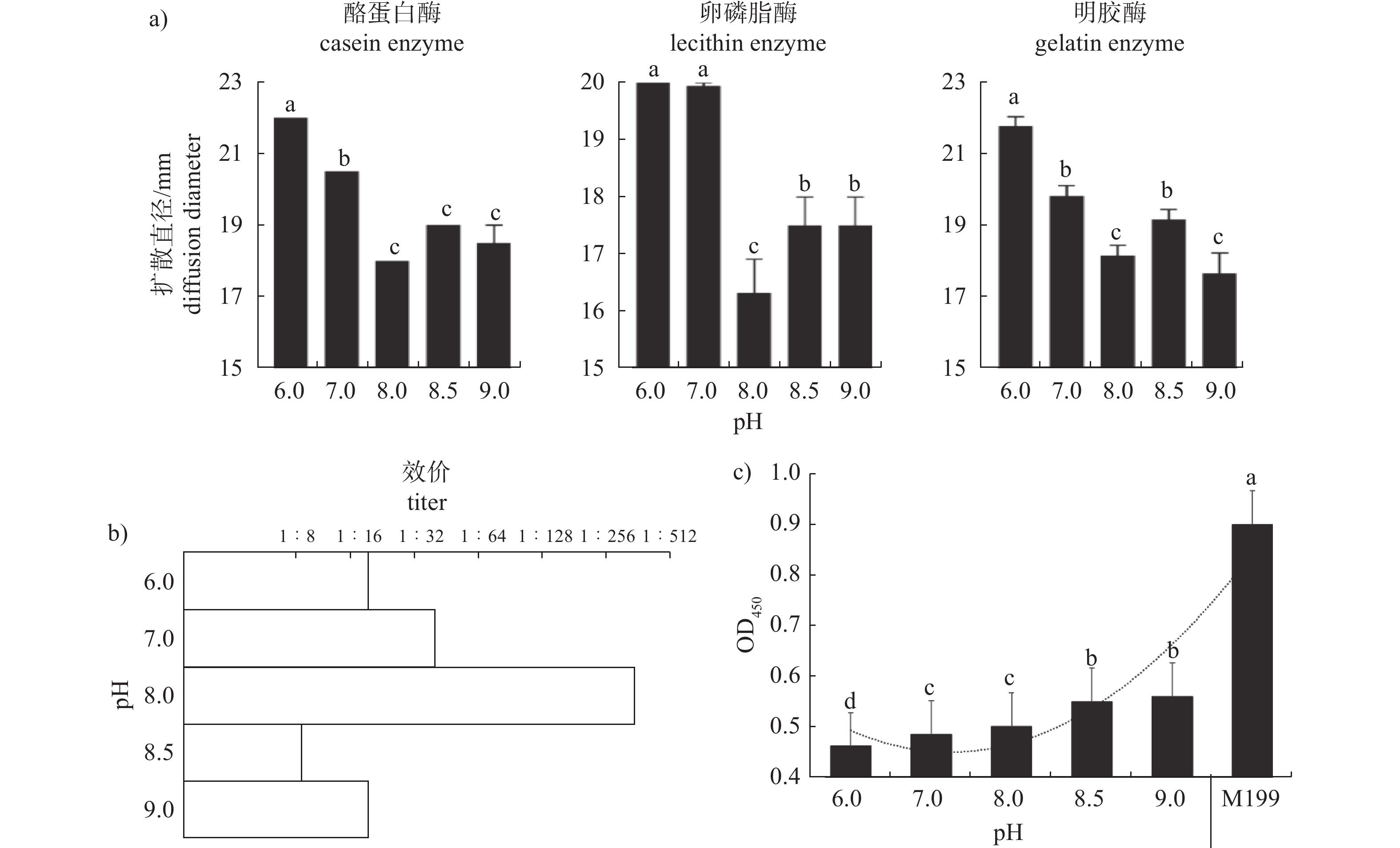
采用琼脂扩散法[14]检测ECPs的酪蛋白酶、卵磷脂酶和明胶酶活性。ECPs质量浓度为0.25 mg/mL,加样量为30 μL;28 ℃正置培养24 h后分别测量平板透明圈直径。
1.4.3 对ECPs溶血性的影响
采用微量板法检测溶血性[14]。将ECPs质量浓度调至1.0 mg/mL后连续稀释2倍,每孔加入等量1%小鼠红细胞,37 ℃孵育1 h后在4 ℃条件下孵育过夜,以50%以上的红细胞被溶解作为判定标准测定溶血效价(即使50%红细胞溶解的样品最高稀释度)。
1.4.4 对ECPs细胞毒性的影响
参考DHARANEEDHARAN等[15]的方法,用M199培养基(含L丙氨酸-L-谷氨酸)将ECPs质量浓度调整至1.0 mg/mL后,加入铺满鲤上皮瘤细胞(epithelioma papulosum cyprini,EPC)的细胞瓶,以M199培养基为阴性对照,在25 ℃恒温培养箱中静置过夜,采用CCK-8试剂盒检测细胞活性。
1.4.5 对ECPs毒力的影响
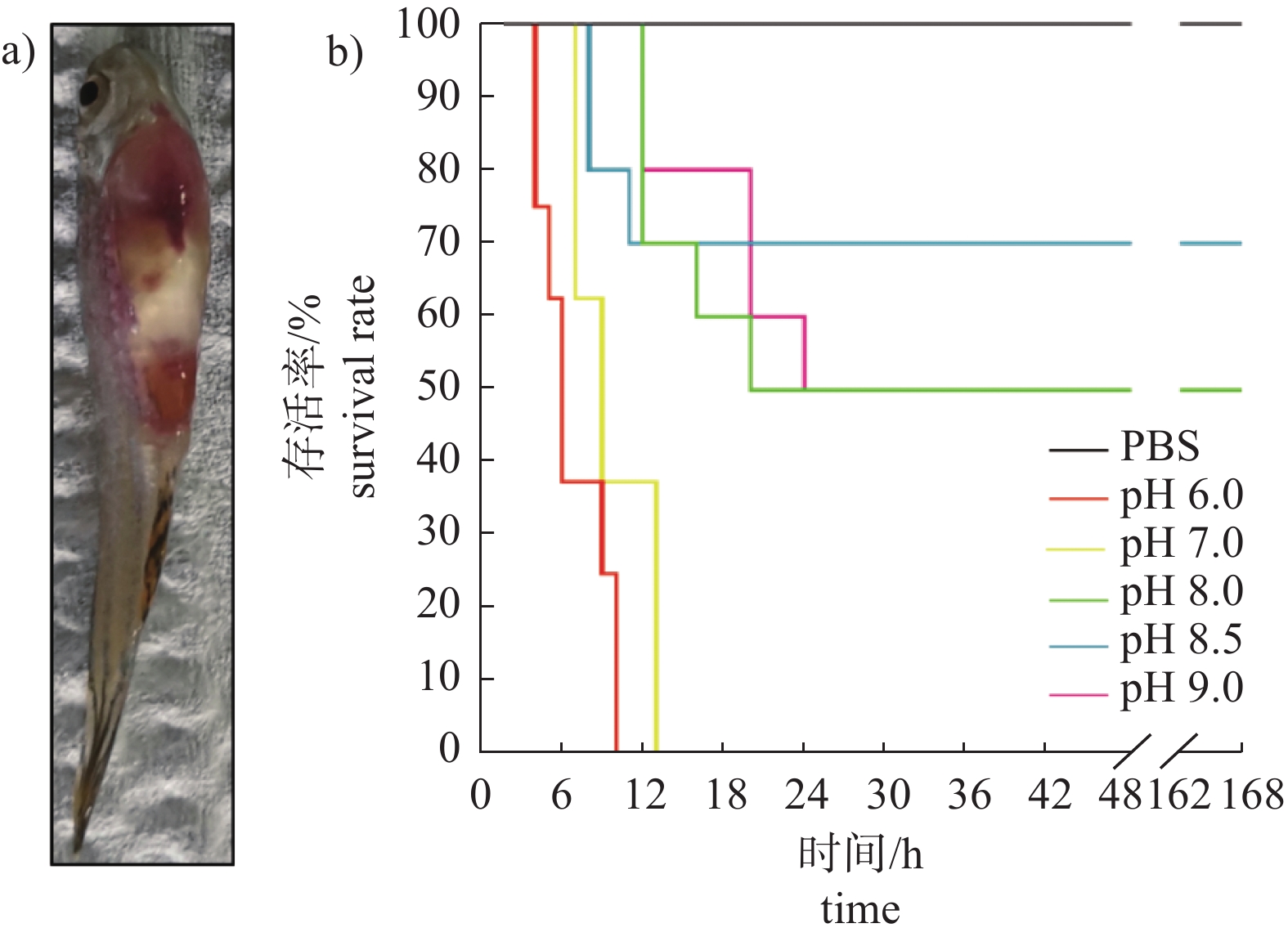
将斑马鱼120尾随机分为6组,每组20尾。试验组分别注射不同pH条件下培养的拟态弧菌ECPs (1.0 mg/mL),每尾腹腔注射50 μL;对照组腹腔注射等量无菌PBS。每隔12 h观察1次,试验期7 d。
1.5 pH对拟态弧菌转录水平的影响
根据感染相关表型与胞外产物特性的研究结果,选择毒力较强(pH 7.0)与毒力较弱(pH 8.5)条件下培养的拟态弧菌进行转录组分析。细菌在28 ℃、130 r/min培养18 h后将OD600值调为1.0,取50 mL提取总RNA后送至上海美吉生物科技有限公司进行转录组测序,提取方法参考JIA等[16],每个处理设置3个生物学重复。使用Bowtie2将读码映射到V. mimicus SCCF01 (登录号:CP016383.1)的参考基因组,差异基因筛选标准为P<0.05,log2FC≥2。采用fasta软件进行数据质控,采用DEseq2进行基因间差异表达分析,所有分析均使用上海美吉生物科技有限公司的I-Sanger 云平台(www.i-sanger.com)进行。
1.6 数据统计与分析
所有数据使用Excel与GraphPad Prism进行统计与处理。
2. 结果与分析
2.1 pH对拟态弧菌感染相关表型的影响
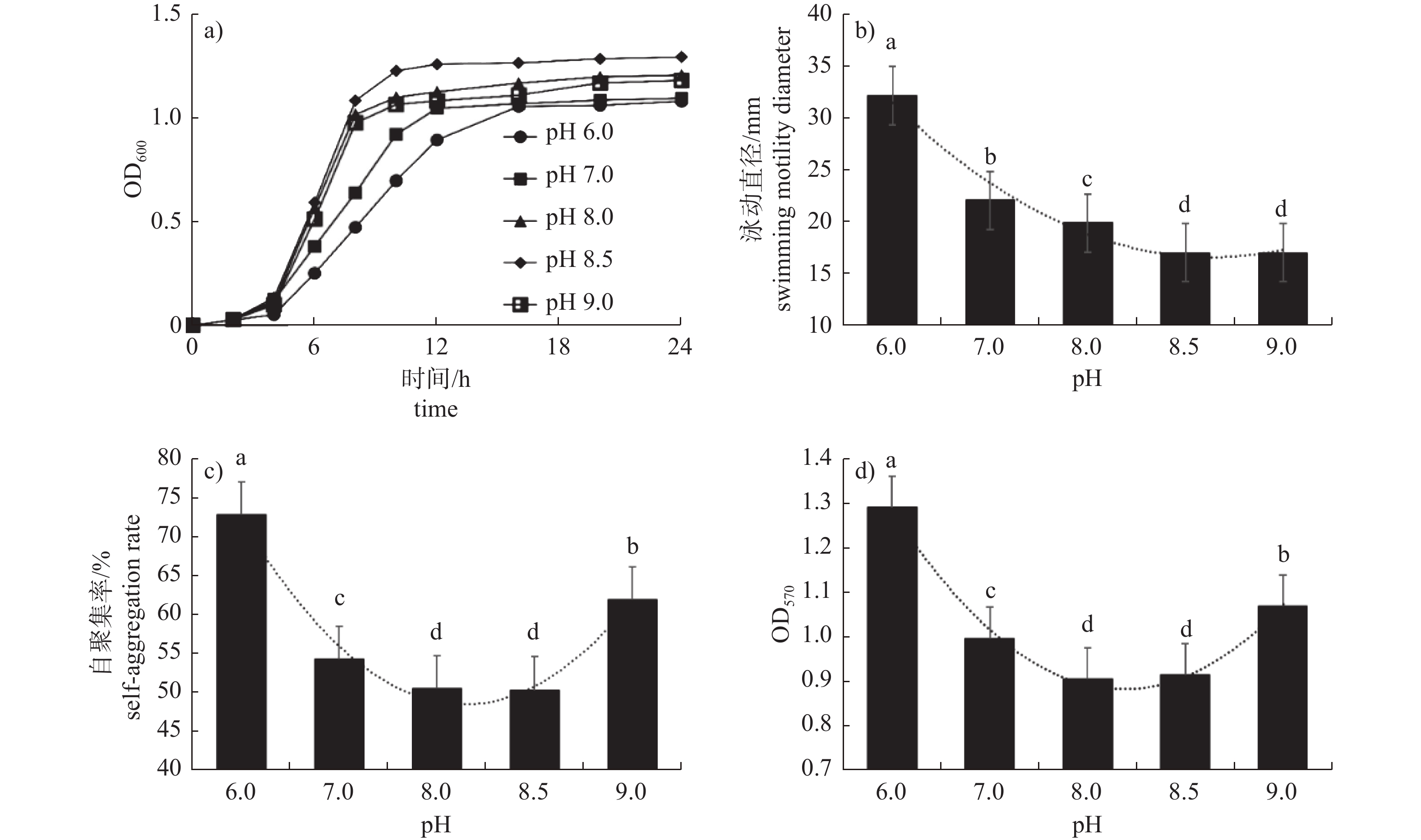
由图1可知:随着pH的升高,拟态弧菌的增殖先增强后减弱,泳动呈减弱趋势,而自聚集和生物膜形成呈先减弱后增强的趋势。环境pH 6.0时,拟态弧菌在增殖12 h后进入平台期,pH 7.0时增殖10 h进入平台期,pH 8.0~9.0均在约8 h进入平台期,pH 8.5时增殖速度最快;pH 6.0时拟态弧菌的泳动、自聚集和生物膜形成均显著强于其他pH (P<0.05),pH 8.5时最弱。
![]() 图 1 不同pH培养后拟态弧菌的感染特性注:a) 增殖,b) 泳动性,c) 自聚集,d) 生物膜形成;不同小写字母表示组间差异显著(P<0.05),下同。Figure 1. Infection characteristics of Vibrio mimicus after cultured in different pHNote: a) proliferation, b) swimming motility, c) self-aggregation, d) biofilm formation; different lowercase letters indicate significant differences among groups (P<0.05), the same as below.
图 1 不同pH培养后拟态弧菌的感染特性注:a) 增殖,b) 泳动性,c) 自聚集,d) 生物膜形成;不同小写字母表示组间差异显著(P<0.05),下同。Figure 1. Infection characteristics of Vibrio mimicus after cultured in different pHNote: a) proliferation, b) swimming motility, c) self-aggregation, d) biofilm formation; different lowercase letters indicate significant differences among groups (P<0.05), the same as below.2.2 pH对拟态弧菌ECPs特性的影响
2.2.1 对ECPs蛋白质量浓度的影响
由表1可知:pH 6.0时ECPs蛋白质量浓度最高,pH 7.0时ECPs蛋白质量浓度次之,pH 9.0时提取的ECPs蛋白质量浓度最低。
表 1 不同pH培养拟态弧菌后提取的ECPs质量浓度Table 1. Mass concentration of extracellular products of Vibrio mimicus in different pH culturespH c/(mg·mL−1) 6.0 3.11±0.11 a 7.0 2.82±0.10 a 8.0 2.52±0.15 a 8.5 1.82±0.13 b 9.0 1.72±0.12 b 注:不同小写字母表示组间差异显著 (P<0.05)。
Note: Different lowercase letters indicate significant differences among groups (P<0.05).2.2.2 对ECPs酶活性、溶血性和细胞毒性的影响
由图2可知: 随着pH的升高,ECPs酶活性和溶血性呈波动变化,但细胞毒性呈现逐渐减弱的趋势。pH 6.0时,ECPs酶活性强于其他pH处理,但溶血性较弱,效价为1∶16;pH 7.0 时,ECPs酶活性仅次于pH 6.0处理,卵磷脂酶活性与pH 6.0时无显著差异,溶血效价为1∶32;pH 8.0时,ECPs酶活性最弱,但溶血性最强,效价为1∶256;pH 8.5时溶血性最弱,溶血效价为1∶8;pH 9.0时,明胶酶活性最弱,溶血效价为1∶16。各pH条件下培养拟态弧菌的ECPs对EPC都具有明显的细胞毒性,与M199组的OD450值相比差异显著(P<0.05),且随pH的升高OD450值增大,ECPs细胞毒性逐渐减弱。
2.2.3 对ECPs毒力的影响
由图3可知:pH对拟态弧菌ECPs的毒力具有明显的影响,pH 6.0与pH 7.0 ECPs攻毒斑马鱼后6 h开始死亡,死亡鱼均表现为胸鳍基部、腹部出血,肛门红肿,攻毒后12 h死亡率为100%;pH 8.5 ECPs攻毒后8 h开始死亡,12 h后停止死亡,试验结束时死亡率为30%;pH 8.0与pH 9.0 ECPs攻毒12 h后开始死亡,试验结束时死亡率为50%。
2.3 pH对拟态弧菌转录水平的影响
2.3.1 转录组原始数据质控评估
vm7-1、vm7-2、vm7-3、vm8.5-1、vm8.5-2和vm8.5-3转录组数据的测序产量如表2所示,其碱基数均大于2 G。Q20均大于98%,表明本试验测序数据质量合格,可以进一步对基因表达情况进行定量分析。
表 2 pH 7.0和pH 8.5处理拟态弧菌转录组测序数据质量评估Table 2. Quality assessment of V. mimicus transcriptome sequencing data at pH 7.0 and pH 8.5样品
samplemRNA长度/bp
length of mRNA原始数据质量评估/%
quality assessment of raw data测序产量/bp
clean reads待分析碱基数
clean basesQ20 Q30 vm7-1 124.6 98.77 96.26 27165408 3385676131 vm7-2 123.5 98.88 96.55 26983066 3332774405 vm7-3 125.0 98.61 95.85 27141086 3391786776 vm8.5-1 117.5 98.90 96.58 28449398 3343764073 vm8.5-2 118.8 98.82 96.37 28241014 3354626322 vm8.5-3 121.3 98.76 96.26 27863870 3378879868 2.3.2 差异表达基因统计与KEGG富集分析
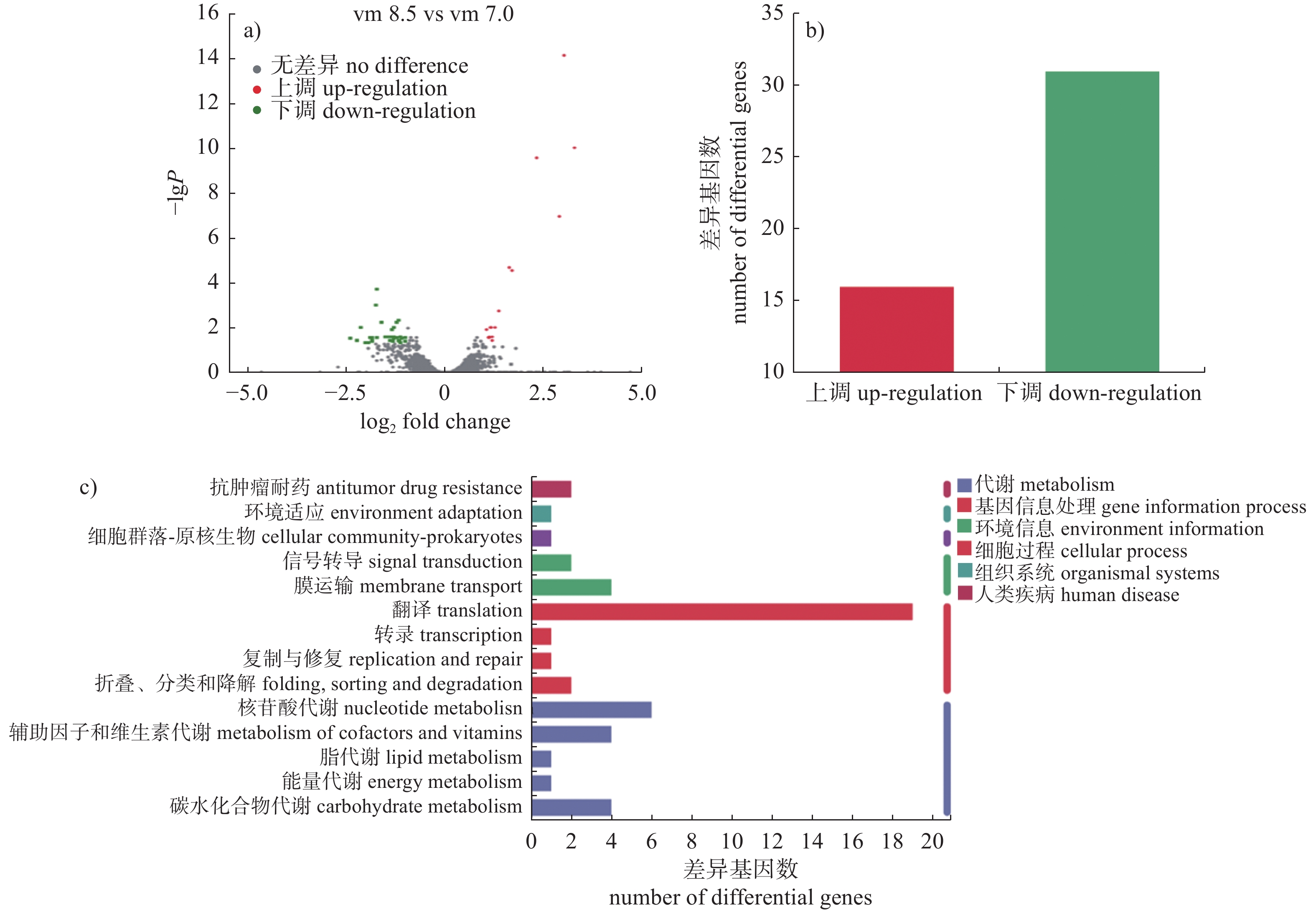
分析结果显示:pH 8.5与pH 7.0培养的拟态弧菌转录结果相比,共有47个基因表达有显著性差异(图4a),其中包括16个上调基因和31个下调基因(图4b)。根据差异基因的 KO编号将基因分别注释到KEGG通路,结果(图4c)显示:差异基因主要富集于碳水化合物代谢、核苷酸代谢、膜转运和翻译等过程。
2.3.3 基因表达量差异分析
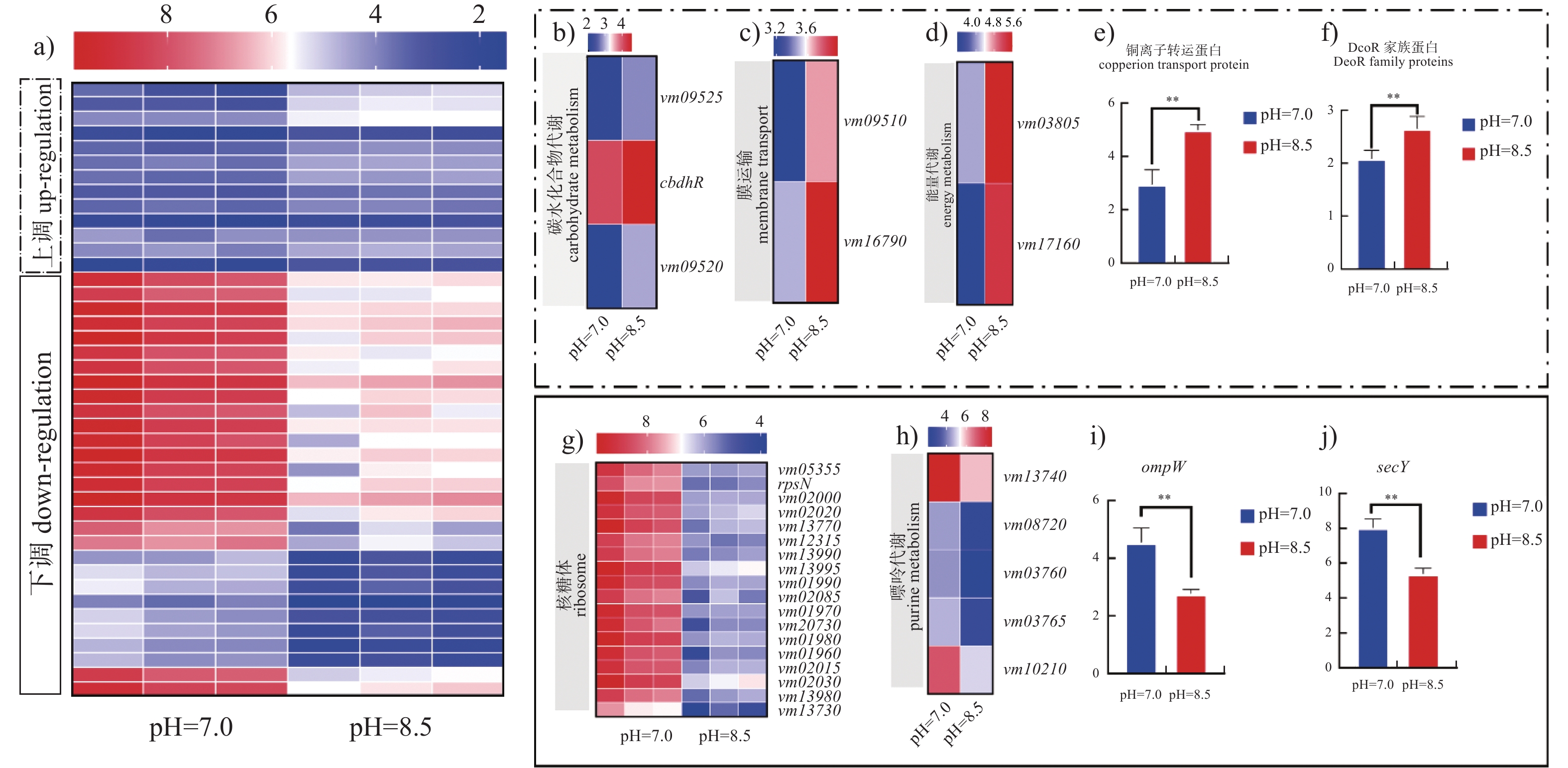
分析差异基因表达显示:与pH 7.0处理相比,拟态弧菌pH 8.5处理后部分基因表达上调,但更多的基因表达显著下调(图5a)。pH 8.5时,拟态弧菌参与碳水化合物代谢和能量代谢过程中的糖酵解和氧化磷酸化相关基因(3-磷酸甘油脱氢酶vm16550和pdhR等)以及参与转运调控的DeoR家族蛋白和铜离子转运蛋白等基因表达显著上调(P<0.05) (图5b~f);而参与核苷酸代谢和辅助因子与维生素代谢的腺苷酸激酶vm10210、磷酸核糖基甘氨酰胺甲酰基转移酶vm03760以及参与翻译和转运过程的核糖体装配基因rspN、外膜蛋白ompW和前蛋白转位酶亚基secY等基因表达显著下调(P<0.05) (图5g~j)。
![]() 图 5 差异基因表达量分析注:a) 显著差异基因整体表达情况;b)~f) 显著上调基因;g)~j) 显著下调基因。“**”表示差异极显著(P<0.0l)。Figure 5. Analysis of differential gene expression levelsNote: a) the overall expression of significantly different genes; b)-f) significantly up-regulated genes; g)-j) significantly down-regulated gene; “**” indicates extremely significant difference (P<0.0l).
图 5 差异基因表达量分析注:a) 显著差异基因整体表达情况;b)~f) 显著上调基因;g)~j) 显著下调基因。“**”表示差异极显著(P<0.0l)。Figure 5. Analysis of differential gene expression levelsNote: a) the overall expression of significantly different genes; b)-f) significantly up-regulated genes; g)-j) significantly down-regulated gene; “**” indicates extremely significant difference (P<0.0l).3. 讨论
环境变化影响微生物的数量与分布,研究表明:创伤弧菌、副溶血弧菌和霍乱弧菌等致病性弧菌的数量与生存环境变化密切相关,低温、低盐和低磷酸盐环境中的弧菌丰度较低、数量较少;水温在20 ℃以上、环境中离子强度、盐度和酸碱度适宜的环境中弧菌种类丰富[17]。不同弧菌最适生长pH不同,霍乱弧菌为pH 7.1,而副溶血弧菌为pH 8.0[18],本研究pH在6.0~8.5范围内,随着pH升高,拟态弧菌增殖力逐渐上升,pH 8.5时增殖力最强。一般认为细菌致病性取决于细菌的入侵数量与毒力,虽然本研究未开展pH对拟态弧菌黏附和入侵力影响的测定,但发现在pH 8.0~8.5条件下,拟态弧菌的ECPs蛋白质量浓度、酶活性及其对斑马鱼的毒力相对较低。ECPs是细菌重要的毒力因子,其中的多糖等物质可帮助病原黏附在宿主表面,蛋白酶等破坏组织和器官等完整性,从而协助病原吸附和入侵,并在宿主体内繁殖、免疫逃避和扩散[19]。本研究表明:不同pH条件下,拟态弧菌产生ECPs特性具有明显差异,尤其是pH 8.5时其毒力最弱,这可以作为养殖生产中开展水质pH调控抵御拟态弧菌感染的参考。
生物膜也称为生物被膜,其形成与细菌运动、自聚集和胞外产物分泌等特性有关[20]。本研究中,随着pH升高,拟态弧菌的运动性、自聚集、ECPs分泌和生物膜形成减弱。生物膜形成既可帮助细菌抵抗不利环境,也可协助细菌免疫逃避造成病原的持续感染,病原可经生物膜定殖于宿主后躲避免疫系统识别[21]。研究表明:环境因子如温度、pH和离子强度等影响细菌生物被膜的形成[22],极端的酸性与碱性环境中形成的生物膜对环境胁迫有更强的抵抗力[23]。本研究中,pH 6.0~8.5时生物膜形成能力随pH升高而减弱,pH 6.0时该能力最强,此时其ECPs分泌量、泳动和自聚集均最强,表明拟态弧菌的感染特性随pH的升高而降低。
代谢是机体产生能量的重要途径,ATP与ADP为其关键底物,且ADP对ATP呈负反馈调节[24]。能量代谢对细菌DNA合成与生长具有重要意义,其代谢中间产物能够调节DNA复制的启动[25]。pH 8.5时,拟态弧菌3-磷酸甘油脱氢酶vm16550和pdhR等参与糖酵解的基因表达显著上调,细菌增殖较快,而参与ADP合成的腺苷酸激酶vm10210和磷酸核糖基甘氨酰胺甲酰基转移酶vm03760等基因表达下调,可能是ADP负反馈调节发挥了作用,但具体原因还需进一步研究。pH 8.5时,拟态弧菌生物膜形成、胞外蛋白酶分泌和ECPs毒力减弱,此时参与多肽翻译的核糖体亚基30S与50S基因以及参与物质跨膜运输的secY和ompW基因表达下调。细菌核糖体亚基30S和50S协作参与多肽链的翻译过程[26];secY蛋白协助蛋白质穿过内膜参与其分泌过程[27],外膜蛋白ompW参与疏水性小分子和铁的转运以及细菌对环境的应激调节[28]。pH 8.5时,拟态弧菌感染相关表型、ECPs蛋白质量浓度及其酶活性等特性减弱可能与相关基因表达下调有关。
4. 结论
pH对拟态弧菌感染相关表型及ECPs特性具有明显影响,pH 8.0~8.5条件下其感染与致病相关特性处于较低水平; pH可能是通过调节拟态弧菌能量代谢与膜运输等过程影响相关生物学特性。
-
表 1 云南野桑蚕mtDNA扩增引物序列
Table 1 Primer sequences of Yunnan Bombyx mandarina mtDNA amplification
引物名称
primer name引物序列 (5′→3′)
primer sequenceGap1 F:GCTGATAGAATTTATGGATC
R:GTCGAAATTAAATGCAAATTATTGCGap2 F:GTACAGTCAAAATACTGCAG
R:GTATCTTGTGTATCAGAGGap3 F:CTCTGATACACAAGATAC-
R:GTGCCAGCAGTTGCGGTTATACATR F:CCGCAACTGCTGGCAC
R:GAGGTATGAGCCC表 2 不同鳞翅目昆虫mtDNA序列信息
Table 2 Information of mtDNA sequence from various Lepidoptera insects
名称
name登录号
accession number基因组长度/bp
genome length名称
name登录号
accession number基因组长度/bp
genome length祥云野桑蚕
Bombyx mandarina XiangyunMZ054176 15 688 彭山野桑蚕
Bombyx mandarina PengshanGU966616.1 15 682 青州野桑蚕
Bombyx mandarina QingzhouFJ384796.1 15 717 洪雅野桑蚕
Bombyx mandarina HongyaGU966606.1 15 682 海阳野桑
Bombyx mandarina HaiyangMK246423.1 15 768 澧县野桑蚕
Bombyx mandarina LixianGU966592.1 15 682 沈阳野桑蚕
Bombyx mandarina ShenyangMG604734.1 15 682 荣昌野桑蚕
Bombyx mandarina RongchangGU966629.1 15 682 南阳野桑蚕
Bombyx mandarina NanyangKT589976.1 15 682 盐城野桑蚕
Bombyx mandarina YanchengGU966598.1 15 682 石泉野桑蚕
Bombyx mandarina ShiquanMN400656.1 15 662 苏州野桑蚕
Bombyx mandarina SuzhouGU966619.1 15 682 安康1野桑蚕
Bombyx mandarina Ankang1AY301620.2 15 682 宜昌野桑蚕
Bombyx mandarina YichangGU966627.1 15 682 安康2野桑蚕
Bombyx mandarina Ankang2GU966591.1 15 682 日本野桑蚕
Bombyx mandarina JapanGU966593.1 15 928 资阳野桑蚕
Bombyx mandarina ZiyangGU966621.1 15 682 家蚕 (C108)
Bombyx mori C108GU966630.1 15 656 南充野桑蚕
Bombyx mandarina NanchongGU966597.1 15 682 家蚕 (夏芳)
Bombyx mori XiafangAY048187.1 15 664 泸州野桑蚕
Bombyx mandarina LuzhouGU966631.1 15 682 柞蚕 Antheraea pernyi AY242996.2 15 566 表 3 云南野桑蚕mtDNA基因分布与注释
Table 3 Gene distribution and annotation of the Yunnan B. mandarina mtDNA
基因或区域
gene or region编码链
coding strand位置/nt
position长度/bp
size反密码子
anticodon起始密码子
start codon终止密码子
stop codon基因间隔/bp
intergenic lengthcox3 J 1~789 789 ATG TAA 2 tRNAGly J 792~857 66 TCC 3 nad3 J 861~1 211 351 ATA TAA 53 tRNAAla J 1 265~1 332 68 TGC 55 tRNAArg J 1 388~1 451 64 TCG 1 tRNAAsn J 1 453~1 519 67 GTT 0 tRNASer (AGN) J 1 520~1 586 67 GCT 13 tRNAGlu J 1 600~1662 63 TTC −1 tRNAPhe N 1 662~1728 67 CTT 4 nad5 N 1 733~3 451 1 719 ATT TAA 21 tRNAHis N 3 473~3 539 67 CAC 54 nad4 N 3 594~4 934 1 341 ATG TAA −1 nad4L N 4 934~5 224 291 ATG TAA 4 tRNAThr J 5 229~5 293 65 TGT 1 tRNAPro N 5 294~5 359 66 ACC 2 nad6 J 5 362~5 892 531 ATT TAA 48 cytb J 5 941~7 098 1 158 ATA TAA −1 tRNASer (UCN) J 7 098~7 163 66 TGA 25 nad1 N 7 189~8 127 939 ATT TAA 0 tRNALeu (CUN) N 8 128~8 198 71 ATC 30 LrRNA N 8 229~9 594 1 366 0 tRNAVal N 9 595~9 669 75 ATG 1 SrRNA N 9 671~10 453 783 0 A+T丰富区
A+T rich region10 454~10 938 485 0 tRNAMet J 10 939~11 002 64 CAT 0 tRNAIle J 11 003~11 068 66 GAT −3 tRNAGln N 11 066~11 134 69 AAC 47 nad2 J 11 182~12 204 1 023 ATA TAA 5 tRNATrp J 12 210~12 279 70 TCA −7 tRNACys N 12 273~12 337 65 CGT 9 tRNATyr N 12 347~12 408 62 CAT 18 cox1 J 12 427~13 957 1 531 CGA T(AA) 0 tRNALeu (UUR) J 13 958~14 024 67 TAA 0 cox2 J 14 025~14 706 682 ATG T(AA) 0 tRNALys J 14 707~14 777 71 CTT −2 tRNAAsp J 14 776~14 843 68 GTC 0 atp8 J 14 844~15 005 162 ATA TAA −7 atp6 J 14 999~15 676 678 ATG TAA 12 注:cox. 细胞色素c氧化酶亚基基因,nad. NADH脱氢酶亚基基因,cytb. 细胞色素b基因,LrRNA. 核糖体大亚基RNA基因,SrRNA. 核糖体小亚基RNA基因,atp. ATP合成酶亚基;Gly. 甘氨酸,Ala. 丙氨酸,Arg. 精氨酸,Asn. 天冬酰胺,Ser. 丝氨酸,Glu. 谷氨酸,Phe. 苯丙氨酸,His. 组氨酸,Thr. 苏氨酸,Pro. 脯氨酸,Leu. 亮氨酸,Val. 缬氨酸,Met. 蛋氨酸,Ile. 异亮氨酸,Gln. 谷氨酰胺,Trp. 色氨酸,Cys. 半胱氨酸,Tyr. 酪氨酸,Lys. 赖氨酸,Asp. 天冬氨酸;tRNASer (AGN)、tRNASer (UCN)和tRNALeu (CUN)、tRNALeu (UUR)分别为tRNASer与tRNALeu识别的2种氨基酸;基因间的间隔:正值为间隔碱基数,负值为重叠碱基数,0表示无间隔。
Note: cox. cytochrome oxidase subunits, nad. NADH dehydrogenase subunit gene, cytb. cytochrome b gene, LrRNA. large ribosomal RNA gene, SrRNA. small ribosomal RNA gene, atp. ATPase subunit gene; J. majority-strand; N. minority-strand; Gly. glycine, Ala. alanine, Arg. arginine, Asn. asparagine, Ser. serine, Glu. glutamicacid, Phe. phenylalanine, His. histidine, Thr. threonine, Pro. proline, Leu. leucine, Val. valine, Met. methionine, Ile. isoleucine, Gln. glutarnine, Trp. tryptophan, Cys. cystine, Tyr. tyrosine, Lys. lysine, Asp. aspartic acid; tRNASer (AGN), tRNASer (UCN), tRNALeu (CUN) and tRNALeu (UUR) are the two amino acids corresponding to tRNAser and tRNALeu, respectively; intergenic nucleotides: the positive number indicates interval base pairs between genes, the negative numbers indicates that adjacent genes overlap, zero indicates no interval. -
[1] GOLDSMITH M R, SHIMADA T, ABE H. The genetics and genomics of the silkworm, Bombyx mori[J]. Annual Review of Entomology, 2005, 50(1): 71. DOI: 10.1146/annurev.ento.50.071803.130456.
[2] GUO Y, SHEN Y H, SUN W, et al. Nucleotide diversity and selection signature in the domesticated silkworm, Bombyx mori, and wild silkworm, Bombyx mandarina[J]. Journal of Insect Science, 2011, 11(1): 155. DOI: 10.1673/031.011.15501.
[3] NAKAMURA T, BANNO Y, NAKADA T, et al. Geographic dimorphism of the wild silkworm, Bombyx mandarina, in the chromosome number and the occurrence of a retroposon-like insertion in the arylphorin gene[J]. Genome, 1999, 42(6): 1117. DOI: 10.1139/g99-072.
[4] BANNO Y, NAKAMURA T, NAGASHIMA E, et al. M chromosome of the wild silkworm, Bombyx mandarina (n=27), corresponds to two chromosomes in the domesticated silkworm, Bombyx mori (n=28)[J]. Genome, 2004, 47(1): 96. DOI: 10.1139/g03-112.
[5] HOEH W R, STEWART D T, SUTHERLAND B W, et al. Cytochrome c oxidase sequence comparisons suggest an unusually high rate of mitochondrial DNA evolution in Mytilus (Mollusca: Bivalvia)[J]. Molecular Biology & Evolution, 1996, 13(2): 418. DOI: 10.1093/oxfordjournals.molbev.a025600.
[6] LI D, GUO Y R, SHAO H J, et al. Genetic diversity, molecular phylogeny and selection evidence of the silkworm mitochondria implicated by complete resequencing of 41 genomes[J]. BMC Evolutionary Biology, 2010, 10(1): 81. DOI: 10.1186/1471-2148-10-81.
[7] LU C, YU H S, XIANG Z H. Molecular systematic studies on Chinese mandarina silkworm (Bombyx mandarina M.) and domestic silkworm (Bombyx mori L.)[J]. Agricultural Sciences in China, 2002, 1(3): 349. DOI: 10.1006/jfls.2001.0409.
[8] 杜周和, 刘俊凤, 刘斌彬, 等. 基于amy基因的中国野桑蚕遗传多样性及其与家蚕的系统发育关系[J]. 昆虫学报, 2009, 52(12): 1338. DOI: 10.3321/j.issn:0454-6296.2009.12.008. [9] CHEN D B, ZHANG R S, BIAN H X, et al. Comparative mitochondrial genomes provide new insights into the true wild progenitor and origin of domestic silkworm Bombyx mori[J]. International Journal of Biological Macromolecules, 2019, 131(11): 176. DOI: 10.1016/j.ijbiomac.2019.03.002.
[10] PAN M H, YU Q Y, XIA Y L, et al. Characterization of mitochondrial genome of Chinese wild mulberry silkworm, Bombyx mandarina (Lepidoptera: Bombycidae)[J]. Science in China, 2008, 51(8): 693. DOI: 10.1007/s11427-008-0097-6.
[11] LIU Q N, ZHU B J, DAI L S, et al. The complete mitogenome of Bombyx mori strain Dazao (Lepidoptera: Bombycidae) and comparison with other lepidopteran insect[J]. Genomics, 2013, 101(1): 64. DOI: 10.1016/j.ygeno.2012.10.002.
[12] STEWART J B, BECKENBACH A T. Characterization of mature mitochondrial transcripts in Drosophila, and the implications for the tRNA punctuation model in arthropods[J]. Gene, 2009, 445(1/2): 49. DOI: 10.1016/j.gene.2009.06.006.
[13] KIM J S, KIM M J, CHOI D S, et al. Complete mitochondrial genome of the spotted-wing drosophila, Drosophila suzukii (Diptera: Drosophilidae)[J]. Mitochondrial DNA Part B: Resources, 2016, 1(1): 222. DOI: 10.1080/23802359.2016.1155422.
[14] YAGI T, KATOH T, CHICHVARKHIN A, et al. Molecular phylogeny of butterflies Parnassius glacialis and P. stubbendorfii at various localities in East Asia[J]. Genes & Genetic Systems, 2001, 76(4): 229. DOI: 10.1266/ggs.76.229.
[15] KIM M J, PARK J S, KIM H, et al. Phylogeographic relationships among Bombyx mandarina (Lepidoptera: Bombycidae) populations and their relationships to B. mori inferred from mitochondrial genomes[J]. Biology, 2022, 11(1): 68. DOI: 10.3390/biology11010068.
[16] 段佑云. 家蚕起源于黄河中游中华民族发祥地[J]. 蚕业科学, 1983, 9(1): 50. DOI: 10.13441/j.cnki.cykx.1983.01.011. [17] BANKEVICH A, NURK S, ANTIPOV D, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing[J]. Journal of Computational Biology, 2012, 19(5): 455. DOI: 10.1089/cmb.2012.0021.
[18] 鲁成, 刘运强, 廖顺尧, 等. 家蚕线粒体基因组全序列测定与分析[J]. 农业生物技术学报, 2002, 10(2): 163. DOI: 10.3969/j.issn.1674-7968.2002.02.015. [19] PERNA N T, KOCHER T D. Patterns of nucleotide composition at fourfold degenerate sites of animal mitochondrial genomes[J]. Journal of Molecular Evolution, 1995, 41(3): 353. DOI: 10.1007/BF00186547.
[20] KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology & Evolution, 2016, 33(7): 1870. DOI: 10.1093/molbev/msw054.
[21] CHRIS S, FRANCESCO F, ANDREW B, et al. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J]. Annals of the Entomological Society of America, 1994, 87(6): 651. DOI: 10.1093/aesa/87.6.651.
[22] OKIMOTO R, MACFARLANE J L, CLARY D O, et al. The mitochondrial genomes of two nematodes, Caenorhabditis elegans and Ascaris suum[J]. Genetics, 1992, 130(3): 471. DOI: 10.1093/genetics/130.3.471.
[23] XIANG H, LIU X J, LI M W, et al. The evolutionary road from wild moth to domestic silkworm[J]. Nature Ecology & Evolution, 2018, 2(8): 1268. DOI: 10.1038/s41559-018-0593-4.
[24] 刘彦群, 鲁成. 家蚕的起源与进化研究进展[J]. 蚕业科学, 2018, 44(3): 353. DOI: 10.13441/j.cnki.cykx.2018.03.001. [25] YUKUHIRO K, SEZUTSU H, ITOH M, et al. Significant levels of sequence divergence and gene rearrangements have occurred between the mitochondrial genomes of the wild mulberry silkmoth, Bombyx mandarina, and its close relative, the domesticated silkmoth, Bombyx mori[J]. Molecular Biology & Evolution, 2002, 19(8): 1385. DOI: 10.1093/oxfordjournals.molbev.a004200.
[26] DAI L S, ZHU B J, LIU Q N, et al. Characterization of the complete mitochondrial genome of Bombyx mori strain H9 (Lepidoptera: Bombycidae)[J]. Gene, 2013, 519(2): 326. DOI: 10.1016/j.gene.2013.02.002.
[27] LIU Y Q, LI Y P, PAN M H, et al. The complete mitochondrial genome of the Chinese oak silkmoth, Antheraea pernyi (Lepidoptera: Saturniidae)[J]. Acta Biochimica et Biophysica Sinica, 2008, 40(8): 693. DOI: 10.1111/j.1745-7270.2008.00449.x.
[28] HU X L, CAO G L, XUE R Y, et al. The complete mitogenome and phylogenetic analysis of Bombyx mandarina strain Qingzhou[J]. Molecular Biology Reports, 2010, 37(6): 2599. DOI: 10.1007/s11033-009-9781-2.
[29] SHINMURA Y, OKUYAMA H, KIYOSHI T, et al. The complete mitochondrial genome and genetic distinction of the Taiwanese honeybee, Apis cerana (Hymenoptera: Apidae)[J]. Conservation Genetics Resources, 2017, 9(3): 621. DOI: 10.1007/s12686-017-0879-x.
[30] FENN J D, CAMERON S L, WHITING M F. The complete mitochondrial genome sequence of the Mormon cricket (Anabrus simplex: Tettigoniidae: Orthoptera) and an analysis of control region variability[J]. Insect Molecular Biology, 2007, 16(2): 239. DOI: 10.1111/j.1365-2583.2006.00721.x.
[31] SARASTE M. Structural features of cytochrome oxidase[J]. Quarterly Reviews of Biophysics, 1990, 23(4): 331. DOI: 10.1017/s0033583500005588.
[32] 张永红, 邵榆岚, 苏振国, 等. 基于线粒体CO Ⅰ基因的云南野桑蚕进化分析[J]. 蚕业科学, 2020, 46(6): 716. DOI: 10.13441/j.cnki.cykx.2020.06.008. [33] 孟刚, 彭云武, 楚渠. 不同地域来源野桑蚕基于线粒体CO Ⅰ序列的遗传多样性和系统进化分析[J]. 蚕业科学, 2018, 44(2): 249. DOI: 10.13441/j.cnki.cykx.2018.02.009. [34] LAVROV D V, BROWN W M, BOORE J L. A novel type of RNA editing occurs in the mitochondrial tRNAs of the centipede Lithobius forficatus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(25): 13738. DOI: 10.1073/pnas.250402997.
[35] TAYLOR M F, MCKECHNIE S W, PIERCE N, et al. The lepidopteran mitochondrial control region: structure and evolution[J]. Molecular Biology & Evolution, 1993, 10(6): 1259. DOI: 10.1016/0303-7207(93)90227-B.
[36] ZHANG D X, HEWITT G M. Insect mitochondrial control region: a review of its structure, evolution and usefulness in evolutionary studies[J]. Biochemical Systematics and Ecology, 1997, 25(2): 99. DOI: 10.1016/S0305-1978(96)00042-7.
[37] ARUNKUMAR K P, METTA M, NAGARAJU J. Molecular phylogeny of silkmoths reveals the origin of domesticated silkmoth, Bombyx mori from Chinese Bombyx mandarina and paternal inheritance of Antheraea proylei mitochondrial DNA[J]. Molecular Phylogenetics & Evolution, 2006, 40(2): 419. DOI: 10.1016/j.ympev.2006.02.023.
[38] SAITO S, TAMURA K, ANDAOTSUKA T. Replication origin of mitochondrial DNA in insects[J]. Genetics, 2005, 171(4): 1695. DOI: 10.1534/genetics.105.046243.
[39] YAMAUCHI Y, HOEFFER C, YAMAMOTO A, et al. cDNA and deduced amino acid sequences of apolipophorin-IIIs from Bombyx mori and Bombyx mandarina[J]. Archives of Insect Biochemistry & Physiology, 2000, 43(1): 16. DOI: 10.1002/(SICI)1520-6327(200001)43:1<16::AID-ARCH3>3.0.CO;2-W.
[40] SUN W, YU H S, SHEN Y H, et al. Phylogeny and evolutionary history of the silkworm[J]. Science China Life Sciences, 2012, 55(6): 483. DOI: 10.1007/s11427-012-4334-7.
[41] DENG M, LIAO C Q, CHEN Q, et al. Phylogenetic relationships among Bombycinae (Lepidoptera, Bombycoidea, and Bombycidae) based on mitochondrial genomes[J]. Archives of Insect Biochemistry & Physiology, 2022, 111(1): e21889. DOI: 10.1002/arch.21889.
[42] LI A L, ZHAO Q L, TANG S M, et al. Molecular phylogeny of the domesticated silkworm, Bombyx mori, based on the sequences of mitochondrial cytochrome b genes[J]. Journal of Genetics, 2005, 84(2): 137. DOI: 10.1007/BF02715839.
[43] 陈飞, 王健敏, 孙宝刚, 等. 云南松的地理分布与气候关系[J]. 林业科学研究, 2012, 25(2): 163. DOI: 10.3969/j.issn.1001-1498.2012.02.009. -
期刊类型引用(1)
1. 翟玮,孔祥青,陈钊,丁祝进,葛红星,常志强. 循环率对循环水凡纳滨对虾养殖系统水质和细菌群落组成的影响. 江苏海洋大学学报(自然科学版). 2024(04): 1-8 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载: